クローニング
IMCは、クローニング実験の操作をコンピュータ上で実行できることに特徴があります。この際、特殊なデータは不要で、GenBankやEMBLなどの公的なデータベースから入手できる塩基配列データをそのままクローニングすることが可能です。クローニングは注釈付の配列をそのまま、制限酵素消化、PCRプライマー設計、PCR増幅、ライゲーションを実行できます。結果として得られるクローニング生産物はすべてGenBank/EMBLフォーマットで出力されます。Primer情報は塩基配列上に貼り付けられ保存されるるため、Primerの管理にも有用です。
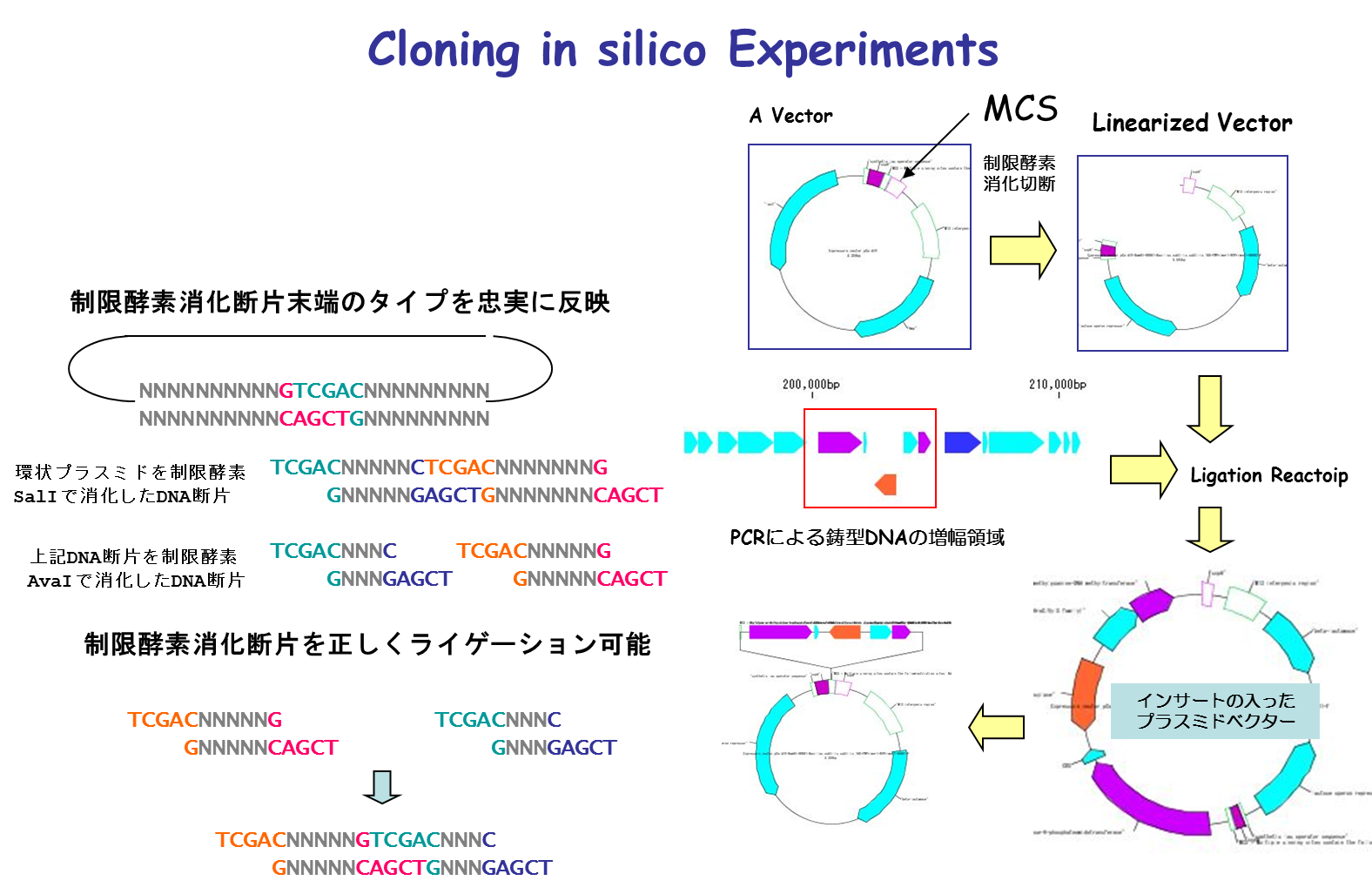
- 実際に切断・ライゲーションできるクローニング機能です。
- インシリコのクローニング実験が可能です。
- 目に見えない分子生物学実験を理解する助けになります。 分子生物学実験の補助やシミュレーションに使用できます。
- 任意のVector/Plasmidをコンストラクトできます。
- Vectorへのインサートチェックに最適な制限酵素をリストアップし、そのゲル電気泳動結果をシミュレート表示します。
- 制限酵素処理(制限酵素地図、制限酵素消化断片生成、インサートチェックに最適な制限酵素候補リスト、その他)
- PCRプライマー設計、
- 特定のフィーチャー上を避けるプライマー設計、
- PCRによる複製(注釈含む)、
- 全遺伝子を増幅する一括プライマー設計、
- プライマー管理 ライゲーション、セルフライゲーション、
- 塩基断片末端形状適合性チェック
- プラスミドマップ作成(インサート領域吹出機能) (レイアウトスタイル対応)
- 注釈を記述したままのクローニング DNA末端への制限酵素認識部位の付加
- DNA末端平滑化(Blunting)、リン酸化(脱リン酸化
- 注釈付き塩基配列の任意領域切り出し
サブカテゴリ
制限酵素の管理
- 制限酵素の管理機能として、以下があります。
- 部分セット選択、
- 種類による絞り込み
- 認識塩基数の区別
- パリンドローム性
- 平滑・突出末端の区別
- DAM/DCMの有無
- 切断箇所数による違い
- 制限酵素リストの別名保存、リスト編集が可能です。
- 酵素新規登録、酵素編集、酵素削除、もできます。
制限酵素認識部位の探索機能
- 制限酵素認識部位の探索機能としては、以下があります。
- 探索領域選択、
- 認識部位検索、
- 認識部位リスト表示、
- 制限酵素地図表示、
- 複数配列からの一括検索
IMCには種々のPCR Primer設計方法があります。
単純に 配列レーン 上の塩基配列や フィーチャーレーン の領域をドラッグした範囲を プライマー として登録する機能
- 配列レーン 上からのプライマー登録
- フィーチャーレーン 上からのプライマー登録
フィーチャーレーン 上のフィーチャーや選択領域の内外を増幅するプライマーを設計する機能
- フィーチャーレーン の選択したフィーチャーを含むあるい含まれる プロダクト を増幅するための プライマー設計
- フィーチャーレーンの選択したゲノム領域を含むあるいは含まれるプロダクトを増幅するためのプライマー設計
一度に多数の領域を増幅するプライマーを一括して設計する機能(Iterate Design 機能つき:設計できなくなる領域がなくなるまで設計を繰り返す機能です)
- Batch PCR Primer Design ゲノム上の指定した フィーチャーキー をもつ全フィーチャーをすべて増幅するための バッチPCRプライマー設計
- Whole Genome Covering PCR Primer Design 全ゲノムあるいは大きなゲノム領域を PCRプロダクト が重なりを持って完全にカバーする PCRプライマー設計
- Sequencing Primer Design キャピラリーシーケンサ のワンリードでカバーできないDNA断片を シーケンシング するためのPCRプライマー設計
参考
以下は、遺伝子クラスター設計 など複数のDNA断片を一度に クローニング するためのPrimer群を一括設計する機能です。デザインからクローニングまでを実行し、クローニングされた生成物をIMCにロードできます。
- In-Fusion Design Ver.1 : In-Fusion Cloning 用のPrimer設計機能
- In-Fusion Design & Build Ver.2 : In-Fusion Cloning 用のPrimer設計機能
- In-Fusion Design & Build Ver.3 : ベクター配列にインサートするDNA断片をドロップして遺伝子クラスターを設計し、ビルドする機能
- Batch Gene Cluster Design & Build : クラスターを構成する遺伝子セットを置き換えて一括多数の遺伝子クラスターを設計し、ビルドする機能
- Combinatorial Design & Build : 遺伝子クラスター を構成する断片を コンビナトリアル に組合せ設計し、ビルドする機能
目的と概要
- ライゲーションは、分子クローニングを実行するために不可欠な実験です。IMCでは、注釈付塩基配列を含む様々な塩基配列同士を実際のクローニング実験と同様に結合して、その産物を生成します。すべての注釈はライゲーション産物にも正しく継承され、かつその産物も元の塩基配列と同様にIMCの多くの機能を使って解析することができます。
機能
- 塩基配列同士をその末端形状が相補的に一致する場合は、ライゲートし、1つの塩基配列にします。
- 最大5断片までの塩基配列をライゲーションできます。
- 3断片以上のライゲーションは、IMCGE以上で実行できます。
- 生成可能な全ライゲーション配列を生成できます。
- Version 7.24以降では、ライゲーション産物の完全相補配列を別な生成物としてカウントしています。
- GenBankやEMBL形式の注釈付塩基配列も同様にライゲーションが可能で、それらの注釈はライゲーション生成物に正しく継承されます。
- 塩基配列の末端同士の形状と配列が相補的に一致する場合は、環状塩基配列を生成します。
- 生成されたライゲーション産物における各断片のインサート方向をチェックするために使用できる制限酵素のリストを表示します。
- またそれらの制限酵素を使用した場合のゲル電気泳動パターンを表示します。
- カレントフォルダーにロードされているすべての塩基塩基配列の末端形状を表示します。
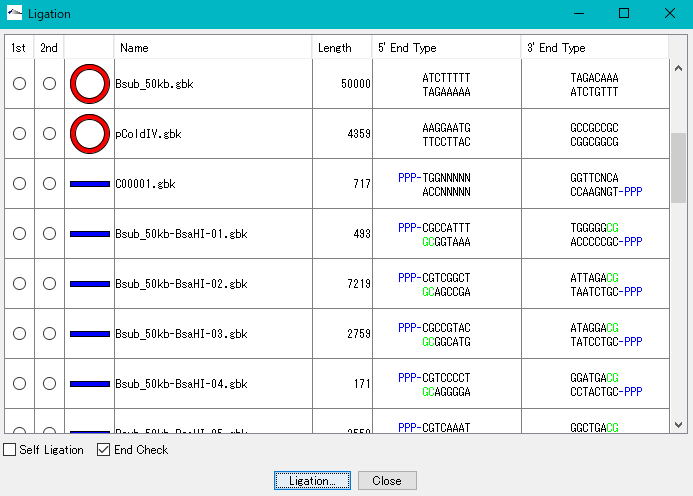
- ライゲーション産物として生成される塩基配列は、元の塩基配列と同様に解析することができます。
制限事項
- 現在、6個以上の塩基配列を連結するライゲーションはできません。
アルゴリズム
- IMC上で制限酵素切断された配列には、末端の状態を示す特殊なFeature KeyとQualifierが生成されます。





